第2部 料紙の構造をさぐる 3 DNAをさぐる★『古文書の科学』全文公開
■本論のPDFはこちら。
■本書全体のPDF・ePubはこちら。
DNAをさぐる
石川隆二
1.植物を識別する
植物の種類を明らかにするためには外部形態をもとに判断することが一般的である。イネ、トウモロコシ、およびダイズを区別するには、まず種子の形態や芽生えの時期に双葉のあることがダイズを見分けることになり、種子の胚乳を形成していることがイネとトウモロコシの共通する特徴である。イネとトウモロコシは大きくなれば葉は茎を取り巻く葉鞘と、その先端に外側に垂れ下がるように葉身が形成される。その葉鞘と葉身の接合部にはカラーと呼ばれる"襟首"のような組織ができる。この組織があるからこそ葉は外側にたれ、光を葉の表面で受けることになる。イネの仲間は大陸を超えて生息しており、各大陸に固有種がみられる。それらの固有種は外見がイネの仲間(イネ属)ではあるが種が異なる。これはわずかな形態の違いとともに交雑した個体がそのあとに種子ができないということを示す。ただ、組織がミクロなレベルになると属や種の識別は非常に困難となる。このようなときにDNAでの判定が行われることで属や種、さらに種のなかの品種の同一性もみることが可能となる。そのために既存の判定材料をもとに利用可能な基盤情報が必要とある。
2.植物ゲノム
植物種の細胞は基本的には細胞壁とそのなかの細胞膜で囲まれており、遺伝情報は細胞のなかのさらに膜に包まれて小区画となる核に存在する。その核の外にも葉緑体ならびにミトコンドリアという小区画があり、それぞれDNAを含んでいる(図1)。
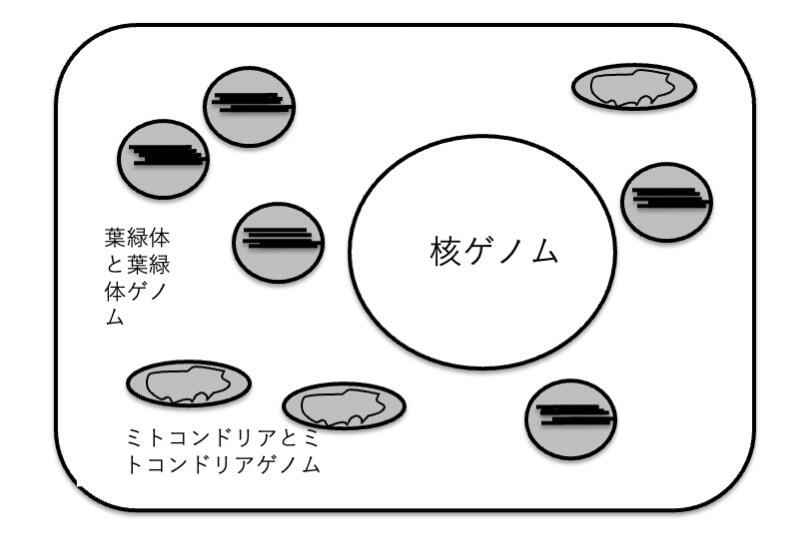
図1 植物の細胞に含まれるゲノム。膜で包まれる核には染色体が納められ,そのDNAは核ゲノムと呼ばれる。細胞質ゲノムは葉緑体とミトコンドリアに含まれる。
これらをゲノムと呼ぶ。植物研究では改良や進化の道筋を明らかにするためにゲノムのDNA配列のすべてを明らかにする研究が進みつつある。それを可能にしたのが次世代シークエンサー(NGS)という技術である。比較的安価になり、高速な処理を可能にする一般的なコンピューターやソフトが入手可能になったことも研究室レベルで利用できることにつながっている。NGSという手法は塩基配列を大量かつ迅速に入手できる塩基配列解析システムの総称である(図2)。150bpのリード(1リードは1回読み込んだデータ)を数億リード得ることで配列データを作成する。そのデータをもとにすでに公表されている塩基配列に対して比較を行うことができる。
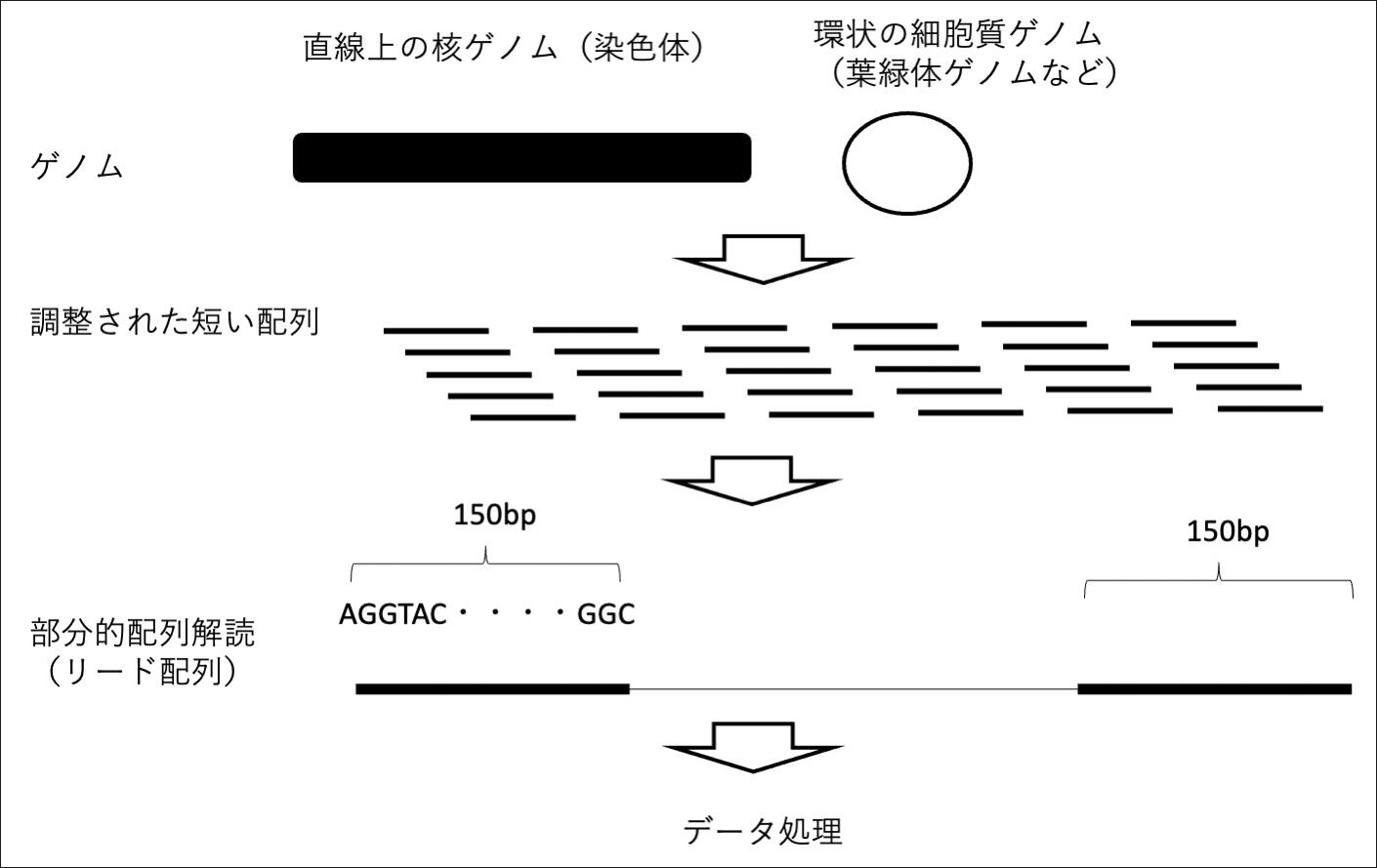
図2 次世代シークエンサー(NGS)によるリード取得の流れ
料紙を形作る成分を研究するときに植物繊維のみならず、その製造過程で混入されるデンプンや残留物である細胞の痕跡、ならびにゲノムの痕跡(短くても種の特徴を有するDNA配列)のあることが期待される。 DNA解析から何か情報が引き出せるとするなら、和紙の材料の地域特異性や、目的ごとに異なる材料が使われていたことなどであろう。それぞれの解析結果が得られるなら料紙復元のために同じ材料を用いることや、書面がしたためられた料紙の生産された場所を特定することも可能になる。
3.どこまでわかるのか
コメ自体が糊などの充填剤として利用されるなら、材料として識別可能なレベルはゲノムの復元率によるものの、二大品種群として知られるインド型か日本型のいずれが用いられたかについては少なくとも知ることができる。そのために葉緑体ゲノムが利用可能である(Ishikawa et al. 2002)。地域ごとに異なる品種が作付けされていたならば、復元したDNA配列により材料のイネ在来種の識別が可能となる。ただし、現時点で入手できるデータは明治以降の在来種の大きな地域分布である。農林水産省では日本のコアコレクションの情報を公開している(Ebana et al. 2008, Tanaka et al. 2020)。この品種をもとにして、単純反復配列(SSRマーカー。ATATATなどの繰り返し構造の変異をもとに品種間識別を行うもの)や、ゲノム上の一塩基多型(SNP)をもとに「現存している在来種のうち供試されたイネと高い類似性を示すもの」を探し出すことが可能である。ここでは同一品種もしくは類似する品種を特定できるが、栽培されていた地域に関する情報は曖昧であるため、それ以上の追跡は困難である。
このように限界はあるものの基盤情報次第で得られる情報量も変化し、蓄積することができる。ただ、イネ科などと大きな分類で素材が利用されるとその途端に科学的な追跡が困難となる。それは分類に用いる"篩い"の網の目が大きくならざるとえず、イネの"篩い"用の編み目ではとらえきれないからである。つまりトウモロコシ、オオムギ、コムギ、サトウキビなどのそれぞれの分類単位を一つの"篩い"では決めることができないからである。かろうじてPS-ID配列が植物種を大きくなら分類できることは報告されている(Nakamura et al. 1997)。
では、そのほかの料紙構成要素ではどうだろう。"カジノキ"もしくは"コウゾ"と呼ばれる植物の繊維は和紙にとっては欠かせないものとなっている。カジノキではどの程度明らかになっているかについては台湾の中国科学アカデミー生物多様性研究センター・研究博物・標本館のChung氏らのグループが東南アジアから日本までの詳細な研究を行っている(Kuo et al. 2022)。彼らのコウゾの歴史によると日本でいうところの"コウゾ"は"ヒメコウゾ"とされ、中国、韓国、日本に分布する植物種であり、かって日本では単に"コウゾ"と呼ばれていた。江戸時代になり"コウゾ"はBroussonetia kajinokiとB. papyriferaとの雑種を意味する言葉とされるようになった。ただし、B. kajinokiはB. monoicaであることが報告された。B. monoicaはしかしヒメコウゾであり、B. monoicaとB. papyriferaの雑種がB. x kajinokiとなることがわかった。
このような背景のもとゲノムデータベース(https://blast.ncbi.nlm.nih.gov )において2種類のBroussonetiaに関する葉緑体ゲノムが登録されていた。NC_037021(160,903 塩基;bp) B. kazinoki x B. papyrifera , KX828844 (160,239 bp) B. papyriferaである。この2種類も上記の種名の混乱の影響を受けている可能性が一部研究者から指摘されている。しかし、塩基配列の比較をすることで異なる領域を検出することができた。いずれも単純な塩基の繰り返し、短い配列の繰り返し数の違い、数塩基の繰り返し(単純塩基反復、SSR)単純な挿入・欠失(INDEL)による違いであった(表1)。これらをDNAマーカーとして用いることにより、個体間の識別を試みることができる(図3)。
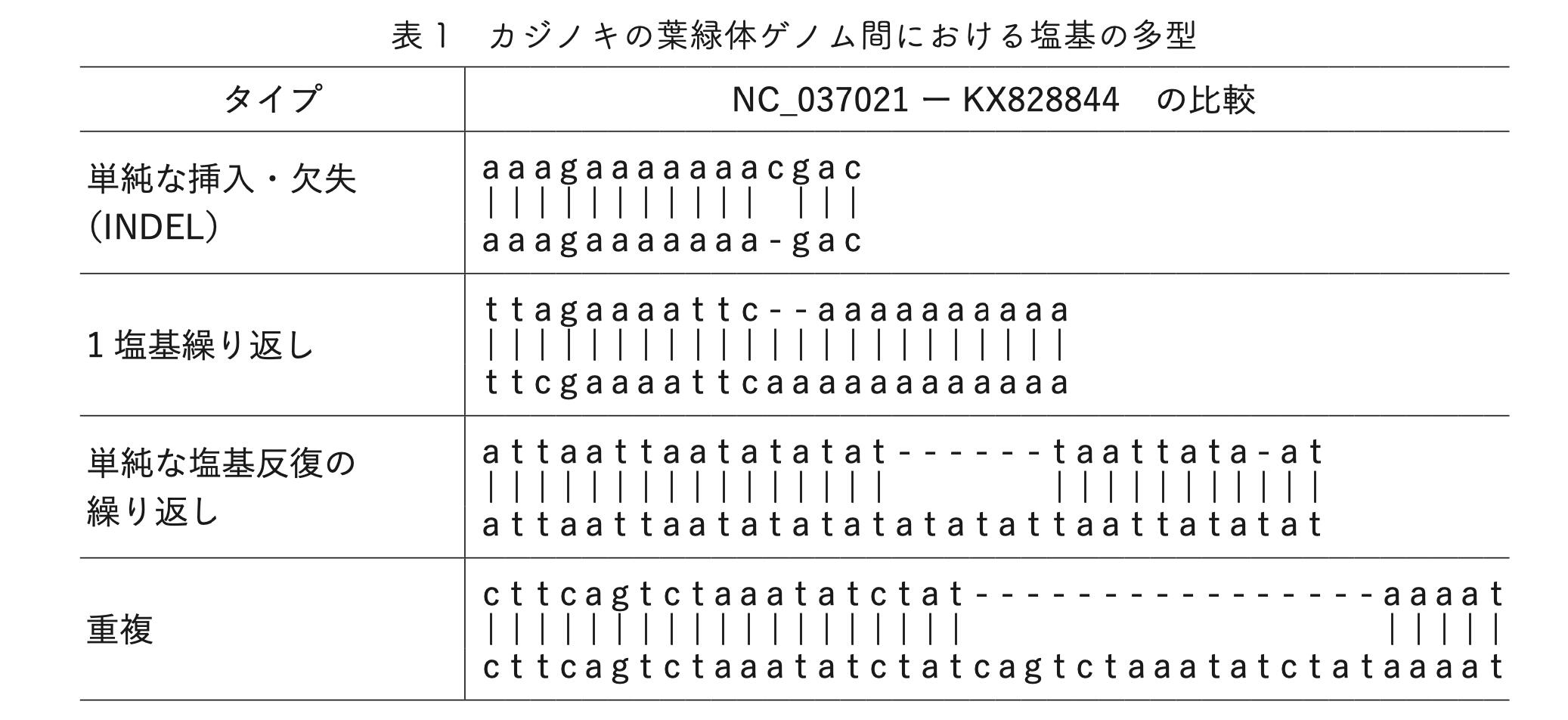
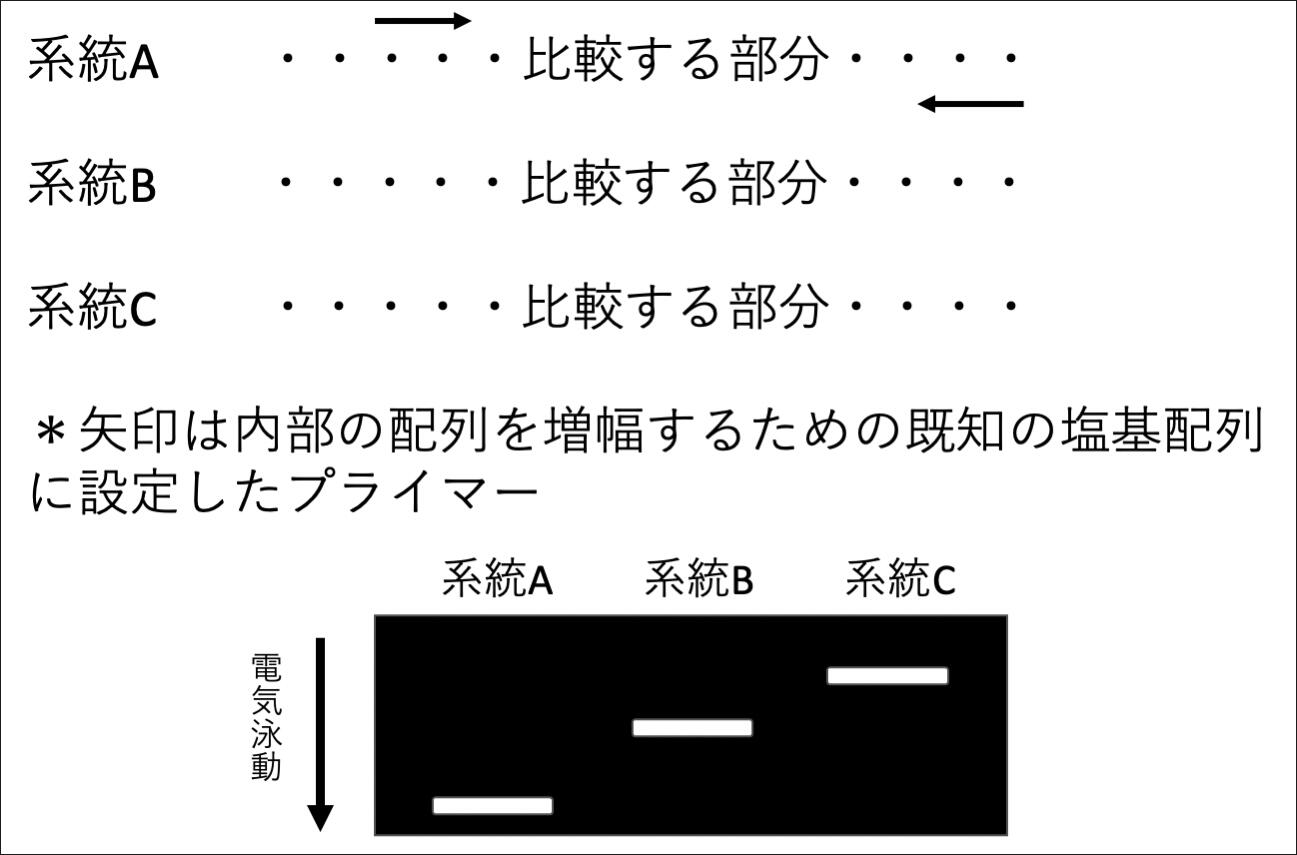
図3 標的配列を増幅するために設定するプライマーと比較手法としての電気泳動
NGSの配列を元に仮想的な長い配列を構成して未知のゲノム配列を再構成(de-novo assembly)することも可能である。カジノキの在来種としてよく知られている。カジノキの既報の葉緑体ゲノムはおよそ16万塩基で構成される。イネの同ゲノムはおよそ12万塩基のため1.2倍大きいことになる。ただ、細胞のなかにあるほかのゲノムである核ならびにミトコンドリアゲノムよりも高いコピー数(データからはおよそ200倍)で存在している。そのため再構成された配列は、それを構成するリードの反復数をもとにほかのゲノムと識別して葉緑体起源であることを見分けることができる。1200万リードを利用した再構成では途中の欠損部分ができてしまったものの、つながった配列においては既知のカジノキ類の配列と相同性が高く、比較に値するデータを取得することができた。このようにコストがかかるものの、初めての植物種をもとに実験をすすめるためのデータを入手することが可能である。得られたデータにおいても、一部の欠損があったものの再構築されたものは葉緑体ゲノムの特徴を示しており、系統間の比較が可能であった。
もう一つのNGSを利用した解析方法はリーシークエンスである。これはChung氏も行っている方法であり、既知のゲノム配列に対して得られたリードを貼り付けることで同じ部分と異なる部分を検出して新たに配列を構築する方法である。こちらはその手順から高速処理が可能である。さらに再構築された配列データから、系統間のDNAの異なる部分(多型、たけい)を予測することができる。これらの予測ができた後に、PCR法と呼ばれる既知の配列間のDNAを増幅する実験手法が適用できる。ここでは20塩基程度の配列にはさまれた200塩基程度の未知の配列を得ることができる。そのため、比較したい系統の葉からのDNA抽出・精製を行ってからの実験となる。図に示したようにプライマーという1本鎖(通常のゲノムは2本鎖)の合成DNAを既知の配列部分に設計する。そのプライマーを合成したのちにPCRを行って、得られた増幅DNAを電気泳動に供試する(図4)。

図4 マーカーで在来種の識別を試みた電気泳動。それぞれのレーンに在来種のPCR産物を泳動している。
ここまで述べた方法は理系の学部学生が取得できる程度の技術であり、今後、応用方法の拡がることが期待される。筆者自身もイネの作物育種学が専門であるが南西諸島の在来カンキツの解析においてはシークヮーサー、ならびに中国の在来作物であるヒシにおいても適用してその成り立ちや多様性程度から進化の経路を推定する成果を得ている。
カジノキにおいてもこのような解析手法において母系列の比較を行うマーカーが作成できる。葉緑体は基本的には母親由来のケースであり、系統を追跡することに利用できる。また、先に述べたように細胞内のコピー数が多いことから、時代を経た料紙など破壊されたDNAについても復元可能性が高い。武藤らは明治時代に備蓄米として残された玄米から比較的高率的にDNAを復元している(Muto et al. 2020)。同じくイネの例では炭化したおよそ5000年前のイネからの復元も行っている(Tanaka et al. 2021)。
4.カジノキの識別
系統間の比較については基盤として現在識別できる在来種間の情報が必要である。そこで初めて過去のDNA情報の比較が可能となる。カジノキの分析では最初に在来種であるアカソ、アオソ、カナメ、ナス 、およびたおりの比較を行った。識別マーカーは上記のゲノム情報から得られたものである(図5)。

図5 ATの8回繰り返し配列をもとにゲノムデータから選抜した多型候補。およそ300万リードから12964配列が見出された。たおりでは14250回、カナメでは14026回見出された。(AT)8は検索に利用した配列での反復数を示す。(AT)14などはATの繰り返しが14回あることを示す。
その結果、Arep1マーカーではナスのみがDNAの増幅がなく、Tand2ではカナメとたおりがやや大きなDNAを示すために泳動が遅れて上部にバンドを示した。Tand3ではカナメとなすが同じ多型を共有しており、ほかと異なることがわかる。これらを情報として表にまとめた(表2)。
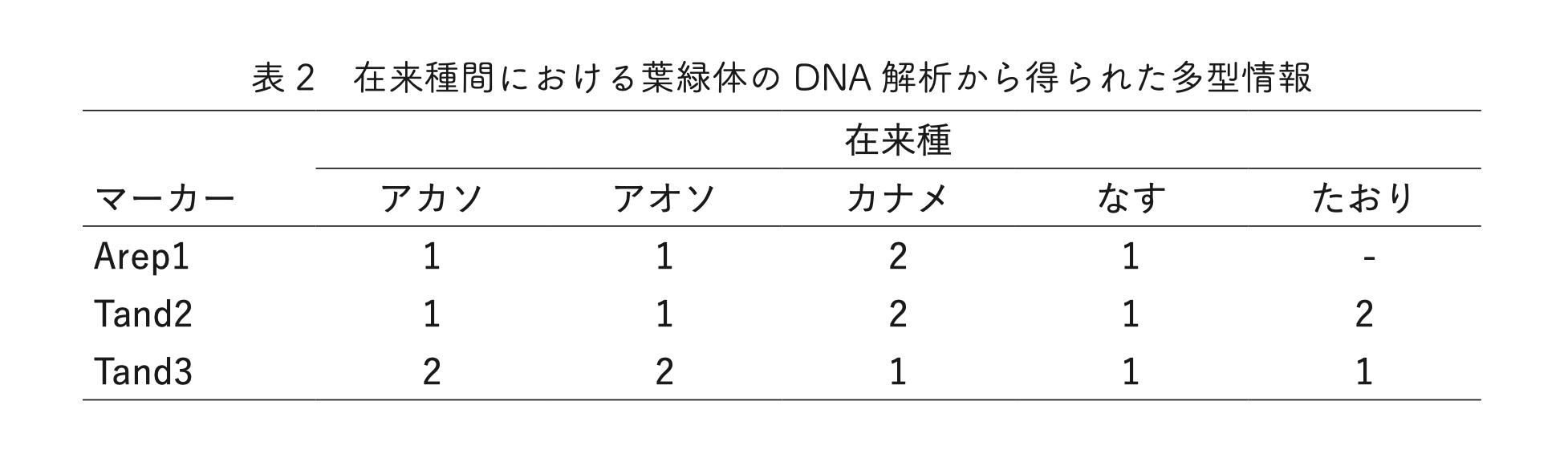
ここではアカソとアオソが同一であることがわかる。ただし、これはすべてのDNA情報を比較したものでないことから、"比較的"高い類似性を示すが、必ずしも"同じ"個体であることをいうものではない。あとで示すMIG-seqなど大量データをもとに、より高い信頼性を持った比較を行うことが必要である。植物分類学的にはより高度な解析データが必要であり、Kuo-fang氏らは供試材料のすべての全葉緑体ゲノムを取得して比較をすすめている。東南アジアから日本にかけての大きな分類情報と詳細な系統関係はこれから明らかにされてくることが期待され、その情報をもとにより高度なマーカー開発も可能である。
母系だけでなく核ゲノムを利用した多型解析は、より精度の高い系統間の比較や個体間の比較を行うことができる。植物の核ゲノムは細胞質ゲノムと呼ばれるミトコンドリアや葉緑体ゲノムと比べて、比較的早い塩基置換が進む。そのため核ゲノムの多型マーカーを供試することで個体間の識別が可能である。上述のイネの在来種の違いが核マーカーにおいて比較されており、品種の識別が可能になっている。いまや日常的に食べている品種の混入の識別でさえも可能である。そのようなときに最も簡単に利用できるマーカーがSSRマーカーである。未知の植物でもNGSによる塩基取得がされていれば簡単に検出できる。ここではATの8回繰り返し構造をデータから入手できた例を示す(図5)。150bpのリードから検索することにより繰り返し構造の両側の配列を抽出した。単純な塩基繰り返し構造の隣接する配列が得られ、その配列をマーカーとして確立することができる。さらに、その隣接配列をほかのNGSデータに検索することで標的の繰り返し構造の違いを検出することも可能であった(表3)。

5.畑のコウゾ
栽培されるコウゾの違いは栽培される畑でみることができる。良質なコウゾを生産する茨城県の大子那須楮を案内していただいたときに異なる外観のコウゾを栽培されていた。そこの栽培されている方は赤楮ならびに黒楮と呼び分けて栽培する畑においてもわけて植えていた(図6)。

図6 大子楮畑の赤楮(左)と黒楮(右)
コウゾは植え付けてからある程度育ったところで上部を刈り取ってコウゾの薄皮を剥いで和紙の原料とする。その後、多年生的なコウゾは親株の根元からひこばえと呼ばれる分枝を伸ばし始める。地下茎からも伸びることがあり、垣根を潜って下から畑の外にでることもあるという。そのため赤楮の区画からほかの区画にひょっこりと新たな枝を伸ばすこともあるだろう。このような色の違いでは、ときに白楮と呼ばれる色の薄いものが出現することもあるという。その理由はわからないものの生きている生物である以上、成長の違いによる外観の違いや元の木とは異なる色の変異を示すことがある。では、このような外観の違いはDNAの違いと関連しているのだろうか。一つ一つのマーカーでの類似性は確率が低く、より多くのマーカーやDNAによる識別が高い信頼性のもと比較が行える。
6.MIG-seqによる解析
ここでもゲノム情報の少ない生物種特有の解析方法がある。MIG-seqもNGSを利用した大量の塩基配列を利用した配列解析方法である(Suyama and Matsuki 2015)。以前からSSR間の配列を解析するInter-SSR(ISSR)が生物間の多型を検証する方法として利用されていた。ゲノムの特定の位置に固有のDNAプライマーが設計できなくてもさまざまなSSRを想定して、その間の配列をPCRで得る方法である。これまでは多型についてDNA泳動のパターンにより生物種間や個体間の識別を行っていた(Gupta et al. 1994)。その配列情報をNGSで一括して取得して、内部の配列の多型をもとに識別する方法が開発され、MIG-Seqと呼ばれている。微量のDNAで適用可能であることから蝶の標本、植物の標本、過去のDNAサンプルなどをもとに同手法を適用することが可能である。カジノキ葉は不純物が多くイネと同じようにはきれいな状態のDNAを入手することができない。さらにサンプルも各地の在来種を個人で集めることが困難な場合に各地の研究協力者に採取を依頼して、送付された葉をもとにDNAを抽出することがある。このような場合、新鮮で若い葉を利用できないこともある。葉は古くなれば多くの液胞を含む、内部に多様な有機物を蓄積する。この有機物がきれいなDNAを抽出するための妨げとなる。このようなときにもMIG-seqは適用可能である。ただし、いまだにコストがかかるため大量サンプルの解析には実用的とはいえない。
筆者は葉緑体DNA解析からグループ化したサンプルをもとにMIG-Seqを適用した。その解析結果を図7に示した。供試したサンプルは各地から送付された野生種や和紙生産者の利用しているコウゾである。特徴的な由来はナスと呼ばれる在来種である。茨城県常陸大宮市役所に当時在籍されていた石井氏に案内していただいた同市内の野生コウゾや栽培コウゾである。この近辺のコウゾはナスコウゾという在来種であるとされる。栃木県那須からの船による輸送により、呼称として"那須"という名称が固定したものという。また、あまりに流通したことからほかの県でもナスコウゾを栽培していることがある。実際に同じ株を分譲され栽培しているケースが想定される。MIG-Seqから得られた多型情報をもとに作成した系統樹からは茨城県の畑で栽培されているコウゾと高知県のナスが非常に近い遺伝的関係が示された (図7)。興味深いことに同じく高知県で栽培されているコウゾでカナメとしているものが非常に近い関係性を示している。葉緑体DNAの多型解析からはこれらは異なるものとして識別されている。母系が異なっていても交雑を通して遺伝的構成要素(核のDNA)が交換されることによりこのような事例は説明できる。
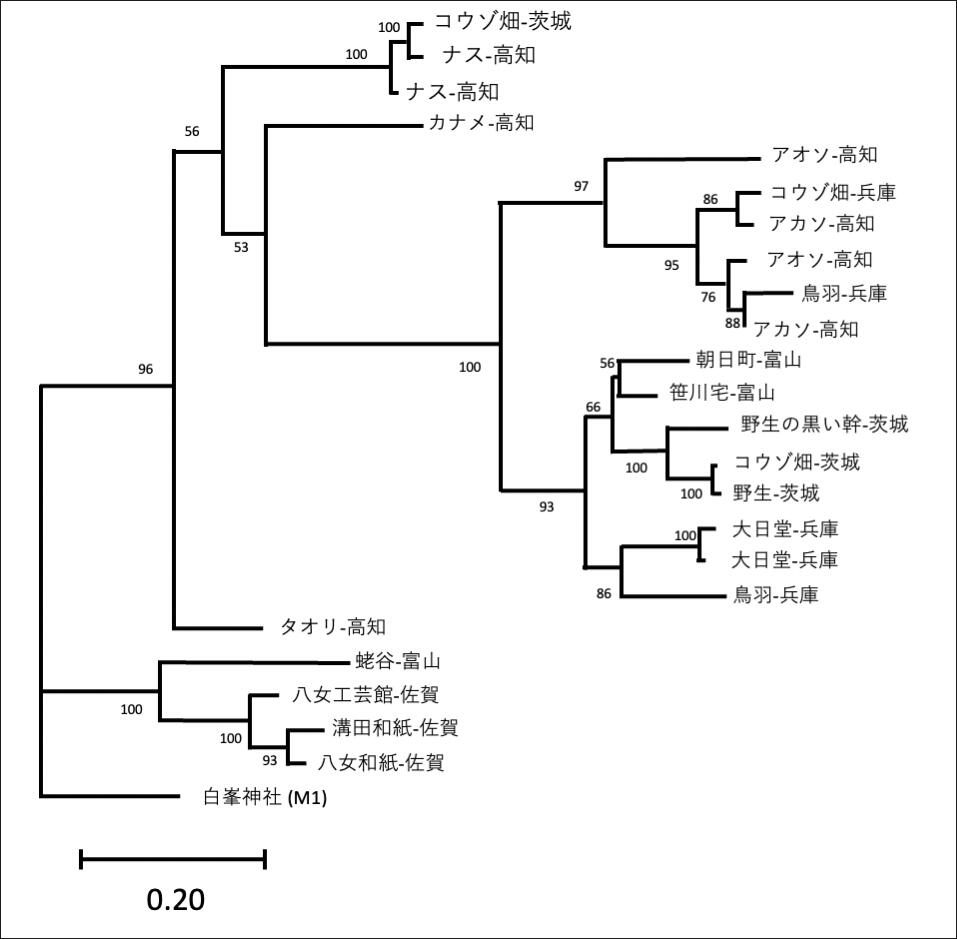
図7 MIG-Seqで得られた情報から行った系統樹解析。数字は信頼性を示し、枝の長さは遺伝的距離を示す。
アオソ、アカソなど同じ高知県の栽培されているものは遺伝的に近い位置に示されたものの同一とは判断できない。この点では、MIG-seqのISSR増幅においてすべての増幅可能領域が増えていなかったときに生じることが考えられる。そのため、どの程度、同一個体を"同一"と判定できるかはやはり、安定したDNAが得られて供試されたかという条件がついていまわる。なお、この類似グループには兵庫県における鳥羽に近い野生種や栽培コウゾも含まれる。同じ種類のコウゾを利用している可能性が指摘できる。
アオソならびにアカソと類似していながらやや異なるグループとしては富山、茨城、兵庫県までの広い地域での野生種が含まれる。一方、タオリは佐賀県八女市の和紙と類似しており、高知県から佐賀県まで広い範囲で類似しているものが利用されていることを示す。なお富山県の蛯谷は野生種として採取されたものである。Chung氏が指摘しているように、日本のコウゾはヒメコウゾであるB. monoicaと栽培種B. papyriferaが交雑したことにより生じたとすると、1回にとどまらず自然界や畑において複数回の交雑が生じた可能性もある。また、鳥がついばんだ種子が広く拡散する過程において、野生種から畑に移し替えて栽培された場合には遺伝的に多様なコウゾが利用されることになる。特に多年生である植物は他個体と交雑した遺伝的雑種(ヘテロ状態)になる可能性が高い。そのまま種子が拡散して利用されることで多様状態が維持されることにもなる。実験では同様な解析を一つの畑で栽培される異なる外観のコウゾにも適用した。赤楮と黒楮である。その結果、外観が異なっても非常に近い類似性を示す赤楮と黒楮もあったものの、黒楮は幅広い遺伝的多様性を示していた。ほかの常陸大宮市の農家の複数の個体も多様性が高かった。そのことからコウゾの栽培は、近年の農業にみられるような集約された農業とは異なる形態で栽培を行っていたことがわかる。
7.神さまのコウゾ
興味深いケースを一つ示す。
京都の白峯神社は蹴鞠の神様としても知られており、Jリーグのプレーヤーも参拝に行くと聞いている。そこには古いコウゾがご神木の一つとして植えられている。もともとコウゾは東南アジアから拡散して朝鮮半島を経由して日本に渡来した可能性もある。紙の製法を含めて渡来人が伝えたときに京都・奈良地域に最初のコウゾの持ち込みを推定することができる。そのため、神社など極めて保存性が高い植生から昔の状態のものが維持されていることも考えられる。そこから徐々に拡散した材料が日本の野生種と交雑を通して多様性を生み出したのだろうか。さらに、熊本県水俣市久木野地区でも製紙産業が盛んなときは、周辺でコウゾ栽培が盛んで、いまでも野生化したコウゾをみることができる。そのように拡散と栽培からの逸脱が起こることで、日本中にコウゾが拡がって野生種と交雑を繰り返していることが想定される。そうなると、DNA解析から料紙の供試材料の地域の特定を行うことは困難である。ただ、同じ性質を有しているだろう個体を現存のサンプルと比較して、補修のための材料として利用可能なものを特定することはできる。
以上のように、DNAの料紙解析への利用には限界もあるものの、今後のDNA解析技術の応用としての可能性を考える材料を提供できるものと思う。さらに、料紙分野を限らず、多様な植物種での応用は今後もコスト的にも適用範囲が拡がるとともに、これまでできなかった方面への応用も期待できるであろう。
参考文献
Chung K-F, Kuo W-H, Hsu Y-H, Li Y-H, Rubite RR, Xu W-B. Molecular recircumscription of Broussonetia (Moraceae) and the identity and taxonomic status of B. kaempferi var. australis. Bot Stud 58:e11, 2017.
Ebana K., Kojima Y., Fukuoka S., Nagamine T., Kawase M. Development of mini core collection of Japanese rice landrace. Breeding Science 58: 281-291, 2008.
Gupta, M., Chyi, Y-S., Romero-Severson J., Owen J. L. Amplification of DNA markers from evolutionarily diverse genomesusing single primers of simple-sequence repeats. Theor. Appl. Genet. 89, 998-1006,1994.
Ishikawa R., Sato Y-I., Tang L. , Nakamura, I. Different maternal origins of Japanese lowland and upland rice populations. Theor. Appl. Genet. 104:976-980, 2002.
Muto C., Tanaka K., Tabuchi H., Kurauchi N., Sato Y-I., Ishikawa R. Genetic diversity of preserved rice seed samples from the Mikawa area, Japan, stored in the Meiji era. Breed. Sci. 70:347-354, 2020.
Suyama Y, Matsuki Y. MIG-seq: an effective PCR based method for genome-wide single-nucleotide polymorphism genotyping using the next generation sequencing platform. Scientific Reports 5: 16963,2015.
Tanaka K., Zhao C., Wang N., Kubota S., Kanehara M., Kamijo N., Ishikawa R., Tasaki H., Kanehara M., Liu B., Chen M., Nakamura S., Udatsu T., Wang C. Classification of archaic rice grains excavated at the Mojiaoshan site within the Liangzhu site complex reveals an Indica and Japonica chloroplast complex. Food Production, Processing and Nutrition 2 :15
Tanaka, N., Shenton M., Kawahara Y., Kumagai M., Sakai H., Kanamori H., Yonemaru J-I., Fukuoka S., Sugimoto K., Ishimoto M., Wu J., Ebana K.. Investigation of the Genetic Diversity of a Rice Core Collection of Japanese Landraces using Whole-Genome Sequencing. Plant and Cell Physiology 61: 2087-2096. Plant and Cell Physiology 61: 2087-2096, 2020.
Kuo W-H., Liu S-H., Chang C-C., Hsieh C-L, Li Y-H, Ito T., Won H., Kokubugata G., Chung K-F. Plastome phylogenomics of Allaeanthus, Broussonetia and Malaisia(Dorstenieae, Moraceae) and the origin of B. x kazinoki. J Plant Res . 135:203-220, 2022.